Open Polymer Prediction Challenge: Analyse des siegreichen Ansatzes
An der NeurIPS Open Polymer Prediction Challenge 2025 nahmen über 2.240 Teams teil, die fünf Polymereigenschaften aus SMILES-Darstellungen vorhersagen wollten: Glasübergangstemperatur (Tg), Wärmeleitfähigkeit (Tc), Dichte (De), fraktioniertes freies Volumen (FFV) und Trägheitsradius (Rg). Wir haben die siegreiche Lösung von James Day analysiert und mehrere wichtige Erkenntnisse identifiziert, die aktuelle Forschungstrends herausfordern und gleichzeitig die anhaltende Effektivität klassischer maschineller Lerntechniken demonstrieren.
Wichtigste Erkenntnisse
Eigenschaftsspezifische Modelle bleiben bei begrenzten Daten überlegen: Trotz des Vorstoßes der Forschungsgemeinschaft in Richtung allgemeiner Stiftungsmodelle haben sich eigenschaftsspezifische Modelle bei der Arbeit mit begrenzten Datensätzen als effektiver erwiesen.
Ensemble-Methoden schneiden weiterhin hervorragend ab: Diese traditionelle Technik des maschinellen Lernens lieferte außergewöhnliche Leistungen, was ihren Wert in modernen Wettbewerben unterstreicht.
Externe Daten erfordern eine sorgfältige Kuratierung: Wie in Die Herausforderungen molekularer Eigenschaftsdatensätze beschrieben, erfordert die Integration externer Datenquellen eine sorgfältige Vorverarbeitung, um Inkonsistenzen und Rauschen zu beseitigen.
BERT für allgemeine Zwecke übertraf die domänenspezifischen Modelle: ModernBERT übertraf die Leistung der chemiespezifischen Modelle, obwohl polyBERT-Einbettungen als wertvolle tabellarische Merkmale beibehalten wurden.
Strategische Auswahl des 3D-Modells: Die siegreiche Lösung verwendete Uni-Mol-2-84M als 3D-Modell. Diese Wahl ist besonders interessant, wenn man bedenkt, dass Praski et al. gezeigt haben, dass Graphen-Transformator-Modelle wie R-MAT bei der Vorhersage von Moleküleigenschaften, insbesondere bei arzneimittelbezogenen Eigenschaften, besser abschneiden. R-MAT-Modelle sind einfach zu implementieren und haben einen geringeren Speicherbedarf, so dass die Wahl von Uni-Mol-2 eine Prüfung wert ist.
Überblick über die Architektur
Der siegreiche Ansatz erstellte eigenschaftsspezifische Vorhersagen unter Verwendung von Ensembles aus ModernBERT-, AutoGluon- und Uni-Mol-2-Modellen durch eine mehrstufige Pipeline:
- Erstes Training auf extern beschrifteten Datensätzen
- BERT-Modell mit einer pseudomarkierten PI1M-Untergruppe neu trainieren
- Umfangreiches Feature Engineering für tabellarische Modelle
- Nachbearbeitungsanpassung für Vorhersagen der Glasübergangstemperatur, um Verteilungsverschiebungen zwischen Trainings- und Leaderboard-Datensätzen zu kompensieren
Daten Strategie
Zusammensetzung und Erweiterung des Datensatzes
Die Modellvalidierung basierte auf einer 5-fachen Kreuzvalidierung unter Verwendung der ursprünglichen Trainingsdaten des Wettbewerbs. Die Trainingsdaten wurden durch externe Datensätze und lokal durchgeführte MD-Simulationen erheblich erweitert. Der Gewinner stellte erhebliche Probleme bei der Datenqualität und eine Verschiebung der Verteilung zwischen den Trainings- und den Leaderboard-Datensätzen fest.
Adressierung der Distributionsverschiebung
Die Untersuchung ergab eine ausgeprägte Verschiebung der Verteilung der Glasübergangstemperatur (Tg) zwischen den Trainings- und Leaderboard-Datensätzen.
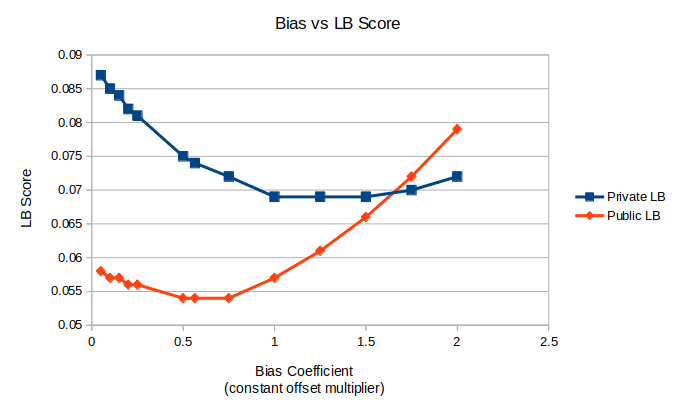 Die untere Grenze (LB) ist die im Wettbewerb verwendete wMAE-Metrik. Der Bias-Koeffizient ist ein Faktor, der mit der Standardabweichung der Glasübergangsvorhersagen multipliziert wird. Dieses Produkt wird dann zu den ursprünglichen Tg-Vorhersagen addiert.
Die untere Grenze (LB) ist die im Wettbewerb verwendete wMAE-Metrik. Der Bias-Koeffizient ist ein Faktor, der mit der Standardabweichung der Glasübergangsvorhersagen multipliziert wird. Dieses Produkt wird dann zu den ursprünglichen Tg-Vorhersagen addiert.
Um diese systematische Verzerrung zu korrigieren, wurden die Vorhersagen nachbearbeitet: submission_df["Tg"] += (submission_df["Tg"].std() * 0.5644)
Externe Datenquellen
Die Lösung umfasste mehrere externe Datensätze:
Diese Datensätze stellten mehrere Herausforderungen dar: zufälliges Label-Rauschen, nicht-lineare Beziehungen zur Grundwahrheit, konstante Verzerrungsfaktoren und Ausreißer aus der Verteilung.
Methodik der Datenbereinigung
Drei allgemeine Strategien wurden auf alle Datensätze angewandt:
Neuskalierung von Bezeichnungen durch isotonische Regression: Ein isotonisches Regressionsmodell transformierte rohe Etiketten, indem es lernte, Ensemble-Vorhersagen aus den ursprünglichen Trainingsdaten vorherzusagen. Dieser Ansatz korrigierte effektiv konstante Verzerrungsfaktoren und nicht-lineare Beziehungen zur Grundwahrheit. Die endgültigen Beschriftungen stellten oft Optuna-abgestimmte gewichtete Durchschnitte von Roh- und umskalierten Werten dar, um eine Überanpassung zu minimieren.
Fehlerbasierte Filterung: Die Vorhersagen des Ensembles wurden verwendet, um Proben zu identifizieren, die einen Fehlerschwellenwert überschritten, und diese wurden verworfen. Die Schwellenwerte wurden als Verhältnis des Probenfehlers zum mittleren absoluten Fehler aus den Ensembletests auf dem Host-Datensatz definiert, um konsistente Schwellenwertbereiche für alle Eigenschaften zu gewährleisten und die Suche nach Optuna-Hyperparametern zu erleichtern.
Stichprobengewichtung: Optuna hat die Gewichtung der Stichproben für jeden Datensatz angepasst, so dass die Modelle minderwertige Trainingsbeispiele angemessen berücksichtigen können.
Datensatzspezifische Interventionen eingeschlossen:
RadonPy: Bei der manuellen Inspektion wurden Ausreißer identifiziert und entfernt, insbesondere Wärmeleitfähigkeitswerte über 0,402, die nicht mit den Ensemble-Vorhersagen übereinstimmten. Optuna hat diese gefilterte Version bei der Abstimmung der Hyperparameter häufig bevorzugt.
MD-Simulationen: Anstatt allgemeine Bereinigungsstrategien anzuwenden, implementierte die Lösung Modellstapelung. Ein Ensemble von 41 XGBoost-Modellen sagte Simulationsergebnisse voraus, und diese Vorhersagen dienten als ergänzende Merkmale für AutoGluon. Dieser Ansatz ermöglichte es den Modellen der zweiten Ebene, beliebige nicht-lineare Beziehungen in potenziell verrauschten Simulationsdaten zu lernen. Die tabellarischen Modelle erzielten eine CV wMAE-Verbesserung von etwa 0,0005 im Vergleich zum vollständigen Ausschluss der Simulationsergebnisse.
Optuna bezog allgemeine Reinigungsstrategien und die RadonPy-Filterregel als Hyperparameter ein und bestätigte deren Wert durch Optimierung.
Strategie zur Deduplizierung
Durch die Erweiterung des Datensatzes wurden doppelte Polymere eingeführt, die durch die Umwandlung von SMILES in die kanonische Form identifiziert wurden. Optuna ermittelte die optimale Gewichtung für Duplikate und entfernte Einträge mit geringerer Gewichtung.
Um zu verhindern, dass der Validierungssatz verloren geht, wurden die Tanimoto-Ähnlichkeitswerte für alle Paare aus Trainings- und Testmonomeren berechnet. Trainingsbeispiele mit Ähnlichkeitswerten von mehr als 0,99 zu einem beliebigen Testmonomer wurden ausgeschlossen, um Beinahe-Duplikate auszuschließen.
MD-Simulation Datengenerierung
Molekulardynamiksimulationen wurden für 1.000 hypothetische Polymere von PI1M in einer vierstufigen Pipeline durchgeführt:
-
Auswahl der Konfiguration: Ein LightGBM-Klassifizierungsmodell sagte die optimale Konfigurationswahl zwischen zwei Strategien voraus:
-
Schnell, aber instabil: psi4's Hartree-Fock Geometrieoptimierung (~1 Stunde pro Polymer, 50% Ausfallrate)
- Langsam und stabil: b97-3c basierte Optimierung (~5 Stunden pro Polymer)
Zu den Klassifizierungsmerkmalen gehörten RDKit-Moleküldeskriptoren, Merkmale des Rückgrats und der Seitenketten sowie Konformer aus der ETKDGv3-Generierung mit MMFFOptimierung.
-
RadonPy Verarbeitung:
-
Ausführung der Bestätigungssuche
- Automatische Einstellung des Polymerisationsgrades, um ~600 Atome pro Kette zu erhalten, unabhängig von der Monomergröße
- Zuweisung von Gebühren
-
Erzeugung amorpher Zellen
-
Gleichgewichts-Simulation: LAMMPS berechnete Gleichgewichtssimulationen mit Einstellungen, die speziell für repräsentative Dichtevorhersagen abgestimmt waren.
-
Extraktion von Eigenschaften: Die benutzerdefinierte Logik schätzt FFV, Dichte, Rg und alle verfügbaren RDKit 3D-Moleküldeskriptoren.
BERT-Implementierung
Modellauswahl
Die Lösung erzielte optimale Ergebnisse mit ModernBERT-base, einem Allzweck-Grundlagenmodell, und nicht mit chemiespezifischen Alternativen. Sowohl ChemBERTa als auch polyBERT schnitten im Vergleich zu ModernBERT-base schlechter ab. Unter den BERT-Varianten für allgemeine Zwecke übertraf ModernBERT Alternativen wie DeBERTa. Angesichts des umfangreichen Trainings von ModernBERT mit Codierungsdaten und der starken Leistung bei Codierungsaufgaben wurde CodeBERT bewertet und schnitt vergleichbar ab. Größere Modelle konnten angesichts der begrenzten verfügbaren Feinabstimmungsdaten keine Verbesserungen erzielen.
Vorschulung auf PI1M
Die Lösung implementierte einen zweistufigen Ansatz für das Vortraining:
Stufe 1: Ein Ensemble aus BERT-, Uni-Mol-, AutoGluon- und D-MPNN-Modellen generierte Eigenschaftsvorhersagen für 50.000 PI1M-Polymere.
Stufe 2: BERT-Modelle wurden mit einer Klassifizierungsaufgabe für paarweise Vergleiche trainiert, bei der vorhergesagt wurde, welches Polymer in jedem Paar höhere oder niedrigere Eigenschaftswerte aufwies. Polymerpaare mit ähnlichen Eigenschaftswerten wurden ausgeschlossen. Das Ziel funktionierte wie ein Multi-Task-Klassifikator, der gleichzeitig die Beziehungen zwischen allen fünf Eigenschaften vorhersagte. Diese zusätzliche Pre-Trainingsphase verbesserte die Leistung gegenüber den Basismodellen von Drittanbietern erheblich.
Feinabstimmung Protokoll
Der Feinabstimmungsprozess folgte den üblichen BERT-Verfahren:
- AdamW Optimierer
- Keine gefrorenen Schichten
- Zeitplan für die Lernrate in einem Zyklus mit linearer Glättung
- Automatische gemischte Präzision
- Beschneidung der Gradientennorm bei 1.0
- Optimal abgestimmte Lernrate, Stapelgröße und Epochenzahl
Die begrenzten Trainingsdaten machten differenzierte Lernraten erforderlich: Die Backbone-Lernrate wurde um eine Größenordnung niedriger angesetzt als die Regressionskopf-Lernrate. Die Wahl von No Frozen Layers hat mich angesichts der begrenzten Trainingsdaten überrascht, aber die differenzierten Lernraten haben wahrscheinlich die Überanpassung und die Instabilität des Trainings abgeschwächt.
Datenerweiterung
Sowohl beim Vortraining als auch bei der Feinabstimmung wurde Chem.MolToSmiles(..., canonical=False, doRandom=True, isomericSmiles=True) verwendet, um 10 nicht-kanonische SMILES pro Molekül zu generieren und so die Trainingsdaten um das Zehnfache zu erweitern. Bei der Inferenz wurden 50 Vorhersagen pro SMILES generiert und mit dem Median als endgültige Vorhersage aggregiert.
Tabellarische Modellierung
Rahmen und Auswahl der Merkmale
AutoGluon diente als Rahmen für die tabellarische Modellierung, wobei Optuna die optimalen Merkmale für jede Eigenschaft auswählte.
Feature Technik
Das Feature-Set umfasste verschiedene molekulare Darstellungen:
Molekulare Deskriptoren und Fingerabdrücke:
- Alle von RDKit unterstützten 2D- und graphbasierten molekularen Deskriptoren
- Morgan Fingerabdrücke
- Fingerabdrücke von Atompaaren
- Topologische Torsionsfingerabdrücke
- MACCS-Tasten
Grafische und strukturelle Merkmale:
- NetworkX-basierte Graph-Funktionen
- Backbone- und Sidechain-Funktionen
- Gasteiger Ladestatistik
- Elementzusammensetzung und Bindungsart-Verhältnisse
Vom Modell abgeleitete Merkmale:
- Vorhersagen von 41 XGBoost-Modellen, die anhand von MD-Simulationsergebnissen trainiert wurden (FFV, Dichte, Rg-Vorhersagen und 3D-Strukturdeskriptoren)
- Einbettungen von polyBERT-Modellen, die auf PI1M vortrainiert wurden
Modellvergleich
Alternative Frameworks, darunter XGBoost, LightGBM und TabM, unterzogen sich einer umfangreichen Abstimmung der Hyperparameter, wobei AutoGluon ein etwa 20-fach höheres Rechenbudget zugewiesen wurde. Trotz dieses zusätzlichen Optimierungsaufwands behielt AutoGluon seine überlegene Leistung bei.
3D Molekulare Modellierung
Uni-Mol 2 84M wurde in erster Linie wegen der Effizienz der Implementierung ausgewählt. Das Modell erforderte kein Feature-Engineering oder benutzerdefinierte Trainingsschleifen, was den Entwicklungsprozess vereinfachte. Bei der Verarbeitung größerer Moleküle mit mehr als 130 Atomen kam es zu Engpässen im GPU-Speicher (24 GB), was sich insbesondere auf die FFV-Trainingsdaten auswirkte. Daher wurde Uni-Mol 2 84M aus dem Ensemble der FFV-Vorhersagen ausgeschlossen.
Erfolglose Annäherungen
Die folgenden Strategien haben die Leistung nicht verbessert:
- Neuronale Graphen-Netzwerke, insbesondere D-MPNN
- GMM-basierte Datenerweiterung aus öffentlichen Notizbüchern
- Chemie-spezifische Einbettungsmodelle
Danksagung
Wir danken James Day für die Bereitstellung des Codes, des Lösungsnotizbuchs und der detaillierten Aufzeichnungen, die diese Analyse ermöglicht haben.
Credits für das Bannerbild: Lone Thomasky & Bits&Bäume / Distorted Lake Trees / Licensed by CC-BY 4.0
Verfasst von

Jetze Schuurmans
Machine Learning Engineer
Jetze is a well-rounded Machine Learning Engineer, who is as comfortable solving Data Science use cases as he is productionizing them in the cloud. His expertise includes: AI4Science, MLOps, and GenAI. As a researcher, he has published papers on: Computer Vision and Natural Language Processing and Machine Learning in general.
Contact


